基因nod32
nod32 升级 时间:2021-02-25 阅读:()
中国细胞生物学学报ChineseJournalofCellBiology2011,33(5):510-519http://www.
cjcb.
orgmiRNA-9基因家族进化分析及其靶基因预测罗丹丹罗玉萍彭娟李思光*(南昌大学生命科学与食品工程学院,南昌330031)摘要microRNAs(miRNA)是真核生物中一类长度约为21~25个核苷酸的非编码小分子RNA,在转录后水平调控基因的表达.
该文在miRBase中搜索后生动物的mir-9基因序列.
47个物种中共搜索到120条mir-9基因序列,说明mir-9基因家族广泛存在于不同物种中.
基因定位显示86%的mir-9基因存在于基因间隔区(IGR),多序列比对发现miR-9基因家族成熟序列的第2位到第8位碱基以及第14位到第18位碱基为保守碱基.
进化分析表明mir-9b和mir-9c可能是此基因家族最早出现的基因形式,即祖先基因.
这些祖先基因经过串联重复、大片段重复、个别碱基的缺失及突变等方式形成了脊椎动物中miR-9-1至miR-9-7数个基因.
分别采用四个miRNA靶基因预测软件对mmu-miR-9的靶基因进行预测,发现miR-9与神经系统发育、心肌系统疾病和跨膜运输系统等密切相关.
该研究为今后进一步研究miRNA调控的神经系统发生和神经细胞生长与分化的机制奠定了基础.
关键词miRNA-9;进化;靶基因收稿日期:2010-10-11接受日期:2011-02-25国家自然科学基金(No.
30971473,No.
31071138)资助项目*通讯作者.
Tel:0791-8304099,E-mail:siguangli@163.
commiRNAs是一类长度为20~28nt的非编码小分子RNA,通过与靶mRNA的互补配对而在转录后水平上负调控基因的表达[1].
miRNA基因以单拷贝、多拷贝和基因簇等多种形式存在于基因组中.
成簇基因之间有的具有同源性,有的无同源性,其中串联基因簇(tandemcluster)中的miRNA一般呈现为协同表达[2,3],暗示着miRNA基因存在复杂的调控机制.
早期的研究认为miRNA基因主要存在于基因间隔区,但近期的研究发现大部分哺乳动物的miRNA基因主要定位在转录单位(transcriptunits,TUs)[2,4],且大多数位于内含子区.
不同物种间的同源miRNA存在高度进化保守性.
如miR-1、miR-34、miR-87在无脊椎动物和脊椎动物中高度保守,序列比对分析发现这些保守序列的碱基差异仅为1-2nt.
Lau等[3]在美丽新小杆线虫(Caenorhabditiselegans)中发现的miRNA,85%在C.
briggsaze基因组序列中具有同源序列.
miRNA在物种间的保守性使其成为探索非编码RNA进化规律的理想分子.
已有研究报道,小鼠和人等哺乳动物中miR-9主要在神经组织中表达[5],并通过调控BAF53a[6]、stathmin[7]等基因的表达参与神经细胞的增殖、迁移等活动.
近期研究显示miR-9还控制着乳腺肿瘤的扩散和发展[8,9],所以,对miR-9的深入研究不仅利于揭示其所介导的调控网络,还将为人类疾病的诊断和治疗提供全新的视野.
本文根据miRNA的进化保守性,通过多序列比对及构建系统进化树的方式分析mir-9基因家族的进化历史.
采用TargetScan、PicTar、microRNA.
org和MicroCosmTargrts等四个miRNA靶基因在线分析软件分析miR-9的靶基因.
通过TheGeneOntology、KyotoEncyclopediaofGenesandGenomes和David软件分析了靶基因的生物学功能,进一步了解miR-9在生物体内的角色及其可能的作用机制.
1材料与方法1.
1mir-9序列的获得从miRBase(http://www.
mirbase.
org/cgi-bin/browse.
pl)中下载部分后生动物的mir-9基因前体序列及成熟序列.
1.
2几种模式生物的mir-9基因定位采用NCBI数据库BLAST(http://blast.
ncbi.
nlm.
罗丹丹等:miRNA-9基因家族进化分析及其靶基因预测511nih.
gov/Blast.
cgi)程序,分别用人、黑猩猩、牛、小鼠、斑马鱼、原鸡和黑腹果蝇等7个模式物种的miRNA前体序列对其基因组数据库进行搜索(BLASTAs-sembledRefSeqGenomes),以确定miR-9在基因组中的位置.
1.
3系统发生分析用ClustalX1.
83软件对所有已搜索物种的mir-9基因的前体序列进行多序列比对分析(multiplese-quencealigment,MSA).
利用MEGA4.
0软件采用基于距离参数的邻接法(Neighbor-joining,NJ),并自展分析(Bootstrap)1000次,构建各个纲目中模式生物的系统进化树.
1.
4靶基因的预测以mmu-miR-9(小鼠miR-9)为研究对象,通过TargetScan(http://www.
targetscan.
org/mmu_50/)、PicTar(http://pictar.
mdc-berlin.
de/cgi-bin/new_PicTar_mouse.
cgispecies=mouse)、microrna.
org(http://www.
ebi.
ac.
uk/enright-srv/microcosm/cgi-bin/targets/v5/search.
pl)及MicroCosmTargrts(http://www.
ebi.
ac.
uk/enright-srv/microcosm/cgi-bin/targets/v5/search.
pl)四个microRNA靶位点分析软件在线分析mmu-miR-9的靶基因,选取四个软件均能预测到的靶基因作为候选靶基因.
通过分析靶基因的功能进一步认识mmu-miR-9的生物学功能.
1.
5靶基因的功能分析利用TheGeneOntology(http://www.
geneontology.
org/)、KyotoEncyclopediaofGenesandGenomes(http://www.
genome.
jp/kegg/)和David(http://david.
abcc.
ncifcrf.
gov/)数据库分析靶基因的生物学功能.
2结果2.
1miR-9基因家族及其在基因组中的定位在miRBase中搜索后生动物的mir-9基因序列,在47个物种中共搜索到120条mir-9基因同源序列.
其中,斑马鱼(Daniorerio)的序列数量最多,共7条,原鸡(Gallusgallus)、大猩猩(Gorillagorilla)、飞蝗(Locustamigratoria)等只有一条或者两条序列.
值得注意的是,人类mir-9在miRBase中存在has-mir-9-1、has-mir-9-2和has-mir-9-3三条序列,而同为人类近亲的大猩猩(Gorillagorilla)和黑猩猩(Pantroglodytes)在miRBase数据库中分别只有一条和两条mir-9序列.
利用has-mir-9的前体序列作为查询序列,采用BLAST程序分别对黑猩猩和大猩猩基因组数据库进行搜索,发现在黑猩猩(Pantroglodytes)中还存在一条miRBase中没有收录的has-mir-9-3的同源序列,在大猩猩(Gorillagorilla)中还存在一条miR-Base没有收录的hsa-mir-9-2的同源序列,mfold折叠证实这些序列都存在miRNA前体典型的二级结构,说明这些物种中还存在目前尚未发现的新的mir-9基因.
以人、黑猩猩、牛、小鼠、斑马鱼、原鸡和黑腹果蝇等7个模式生物mir-9前体序列作为查询序列(QuerySequence),分别对这些生物的基因组DNA进行BLAST搜索,确定各序列在基因组中的位置(表1).
基因定位显示,与其它哺乳动物的miRNA主要定位在内含子区域的情况不同,86%的mir-9基因家族成员位于基因间隔区(intergenicregion,IGR),少数位于蛋白编码基因的内含子区域;mir-9b、mir-9c两个基因位于同一染色体上,并与mir-306、mir-79紧密相连聚集成簇;mir-9a、mir-9b和mir-9c主要出现在较低等的模式生物黑腹果蝇中,而mir-9-1至mir-9-7则主要存在于哺乳类、鸟类、鱼类等脊椎动物中.
mir-9a、mir-9b和mir-9c主要出现在物种进化的早期或低等生物中的现象表明其可能为mir-9家族中较原始的基因.
2.
2miR-9基因家族序列分析将mir-9基因家族的成熟序列进行多序列比对分析(表2),发现为第2位到第8位碱基以及第14位到第18位碱基为保守碱基,说明miR-9基因在不同物种间的高度保守性.
比对结果显示miR-9在其进化过程中可能发生了缺失:有的物种在第9位缺失了碱基A/U/G,有的物种在第23位缺失了碱基A;大多数物种在第12位缺失了碱基U,第24位缺失了碱基U.
第9~11位碱基的相对不保守性可能是因为三个碱基的错配结构更有利于其与靶序列的配对[10].
序列比对发现miR-9与miR-9a的成熟序列相同或只在3'末端相差一个碱基A,但在所有物种的前体序列的相应位置都具有碱基A,所以miR-9与miR-9a可能是相同的miRNA,只是由于3'端的A容易脱落,不同研究者在实验中得到了3'端相差1个A的两种分子.
作为转录后水平调控元件,miRNA成熟序列5'端的第2-8位碱基对于其功能的正常发挥至关重要,被称为"种子序列(seedsequence)"[10].
种子序列主要通过与目标mRNA3'UTR区域的互补配对识别512·研究论文·靶基因,负调控靶基因的表达.
在同源性miRNA中,这段序列通常是保守的,我们的比对结果也证实了这点.
mir-9家族前体序列的同源性分析表明,人与黑猩猩的同源性为100%,人与恒河猴之间的同源性为85%,而人与较低等的两栖类(非洲爪蟾)的同源性约为58%,与果蝇的同源性只有39%.
可见亲缘关系近的物种之间核苷酸序列差异小,亲缘关系远的物种之间核苷酸序列差异大.
仔细分析miR-9的前体序列发现,序列差异主要发生在miR-9前体中成熟序列以外的区域,miRNA成熟序列的区域由于受到选择压力而高度保守.
2.
3miR-9基因家族系统进化树的构建来源于miRBase中的miR-9基因主要分布在哺乳类、鸟类、鱼类、两栖纲和节肢动物门、线虫纲中.
采用MEGA4.
0软件,以这些纲目中的模式物种的mir-9序列构建基于距离参数的邻接法(Neighbor-joining,NJ)[11]构建系统发育进化树(图1).
图1可见,进化树可分为三支:mir-9、mir-9b、mir-9c主要分布在紫色球海胆、Capitellasp.
I(线虫纲)、果蝇(节肢动物门)等无脊椎低等生物中,聚为第一支.
从进化树的分支和距离来看,mir-9、mir-9b、mir-9c出现在进化早期并持续到现在,由此可以推测mir-9、mir-9b、mir-9c可能是mir-9基因家族最早出现的基因形式,即为祖先基因.
而且mir-9b和mir-9c位于同一分支中,说明它们亲缘关系近.
基因定位分析显示mir-9b和mir-9c聚集成簇,暗示它们在进化过程中紧密相连.
第二支主要是分布在节肢动物门的mir-9a及部分环节动物门和线虫门的mir-9和mir-9b.
第三分支主要为人、黑猩猩、鱼类、鸟类等脊椎动物的mir-9基因家族成员.
在这一支中,除同源性较低、亲缘关系较远的文昌鱼(全索亚门)的mir-9单列一小支,其他物种中的mir-9-2、mir-9-5、mir-9-6属同一亚支,而mir-9-1、mir-9-3、mir-9-4、mir-9-7属与之平行进化的另一亚支.
分析发现,miR-9b和miR-9c两个拷贝主要出现在无脊椎动物中,miR-9-1、miR-9-2、miR-9-3、miR-9-4、miR-9-5、miR-9-6和miR-9-7主要出现在脊椎动物中,miR-9和miR-9a在脊椎和无脊椎动物中都有分布.
根据上述的分析和比对结果,可以推测mir-9c作为该基因家族最原始的基因存在于线虫纲,经表1miR-9基因家族在七个物种中的基因位置示意图Table1GenomiclocationofmiRNA-9genefamilyinsevenspecies物种miRNA基因名称染色体基因位置SpeciesNameofmiRNAChromosomeGenepositionHomosapienshas-mir-9-1ChrIIntronofClorf61genehas-mir-9-2chr5IGRbetweenMEF2CandTMEM161Bgenehas-mir-9-3chr15IGRbetweenLoc100288642andLoc254559genePantroglodytesptr-mir-9-1chr1IntronofClorf61geneptr-mir-9-2chr5IGRbetweenMEF2CandTMEM161BgenePtr-mir-9-3chr15IGRbetweenPLOGandLOC453637geneMousemmu-mir-9-1chr3IntronofLoc10042277mmu-mir-9-2chr13IGRbetweenMEF2CandLoc62324genemmu-mir-9-3chr7IGRbetweenPlogandRhcggeneGallusgallusgga-mir-9-1chr21IGRbetweenLOC420111andLOC427091genegga-mir-9-2chrZIGRbetweenLOC769007andLOC426919geneBostaurusbta-mir-9-1chr21IGRbetweenLoc533090andRHCGgenebta-mir-9-2chr7IGRbetweenTMEM1618andMEF2cgeneDanioreriodre-mir-9-1chr16IGRbetweenRhb9andMef2dgenedre-mir-9-2chr10IGRbetweenLOC564822andMef2cagenedre-mir-9-3chr25IGRbetweenLOC560613andRhc9genedre-mir-9-4chr22IGRbetweentmem161aandgata2agenedre-mir-9-5chr5IGRbetweenmef2cbandtmem161bgenedre-mir-9-6chr7IGRbetweenZgc162132andKif7genedre-mir-9-7NOTFoundDrosophilamelanogasterdre-mir-9achr3LIGRbetweenCG93COandSha1genedre-mir-9b/9cchr21IntronofGrpgene,anddre-mir-9b/9cclusteringtogetherwithmir-306、mir-306s、mir-79罗丹丹等:miRNA-9基因家族进化分析及其靶基因预测513表2mir-9基因家族各成熟序列比对Table2Multiplesequencealigmentofmaturesequenceofmir-9genefamilymiRNA名称成熟序列碱基数量miRNA名称成熟序列碱基数量NameofmiRNAMaturesequenceBaseNameofMaturesequenceBasenumebermiRNAnumeberxtr-miR-9-3UCUUUGGUUAU-CUAGCUGUAUGA-22mml-miR-9-3UCUUUGGUUAU-CUAGCUGUAUGA-23ame-miR-9aUCUUUGGUUAU-CUAGCUGUAUGA-23mml-miR-9-2UCUUUGGUUAU-CUAGCUGUAUGA-23bfl-miR-9-2UCUUUGGUUAU-CUAGCUGUAUGA-22mml-miR-9-1UCUUUGGUUAU-CUAGCUGUAUGA-23bfl-miR-9-1UCUUUGGUUAU-CUAGCUGUAUGA-22lla-miR-9UCUUUGGUUAU-CUAGCUGUAUGA-23bmo-miR-9UCUUUGGUUAU-CUAGCUGUAUGA-23age-miR-9UCUUUGGUUAU-CUAGCUGUAUGA-23lgi-miR-9UCUUUGGUUAU-CUAGCUGUAUGA-23mdo-miR-9-2UCUUUGGUUAU-CUAGCUGUAUGA-23cap-miR-9UCUUUGGUUAU-CUAGCUGUAUGA-23mdo-miR-9-1UCUUUGGUUAU-CUAGCUGUAUGA-23tca-miR-9aUCUUUGGUUAU-CUAGCUGUAUGA-23eca-miR-9a-2UCUUUGGUUAU-CUAGCUGUAUGA-23lmi-miR-9aUCUUUGGUUAU-CUAGCUGUAUGA-23eca-miR-9aUCUUUGGUUAU-CUAGCUGUAUGA-23dpu-miR-9UCUUUGGUUAU-CUAGCUGUAUGA-23cfa-miR-9-3UCUUUGGUUAU-CUAGCUGUAUGA-23tni-miR-9-4UCUUUGGUUAU-CUAGCUGUAUGA-23cfa-miR-9-2UCUUUGGUUAU-CUAGCUGUAUGA-23tni-miR-9-3UCUUUGGUUAU-CUAGCUGUAUGA-23cfa-miR-9-1UCUUUGGUUAU-CUAGCUGUAUGA-23tni-miR-9-2UCUUUGGUUAU-CUAGCUGUAUGA-23gga-miR-9-2UCUUUGGUUAU-CUAGCUGUAUGA-23tni-miR-9-1UCUUUGGUUAU-CUAGCUGUAUGA-23gga-miR-9-1UCUUUGGUUAU-CUAGCUGUAUGA-23fru-miR-9-4UCUUUGGUUAU-CUAGCUGUAUGA-23tgu-miR-9-2UCUUUGGUUAU-CUAGCUGUAUGA-23fru-miR-9-3UCUUUGGUUAU-CUAGCUGUAUGA-23tgu-miR-9-1UCUUUGGUUAU-CUAGCUGUAUGA-23fru-miR-9-2UCUUUGGUUAU-CUAGCUGUAUGA-23xtr-miR-9a-2UCUUUGGUUAU-CUAGCUGUAUGA-23fru-miR-9-1UCUUUGGUUAU-CUAGCUGUAUGA-23dya-miR-9a-2UCUUUGGUUAU-CUAGCUGUAUGA-23dre-miR-9-7UCUUUGGUUAU-CUAGCUGUAUGA--23dya-miR-9a-1UCUUUGGUUAU-CUAGCUGUAUGA-23dre-miR-9-6UCUUUGGUUAU-CUAGCUGUAUGA-23aga-miR-9aUCUUUGGUUAU-CUAGCUGUAUGA-23dre-miR-9-5UCUUUGGUUAU-CUAGCUGUAUGA-23dan-miR-9aUCUUUGGUUAU-CUAGCUGUAUGA-23dre-miR-9-4UCUUUGGUUAU-CUAGCUGUAUGA-23dwi-miR-9aUCUUUGGUUAU-CUAGCUGUAUGA-23dre-miR-9-3UCUUUGGUUAU-CUAGCUGUAUGA-23aae-miR-9a-2UCUUUGGUUAU-CUAGCUGUAUGA-23dre-miR-9-2UCUUUGGUUAU-CUAGCUGUAUGA-23aae-miR-9a-1UCUUUGGUUAU-CUAGCUGUAUGA-23dre-miR-9-1UCUUUGGUUAU-CUAGCUGUAUGA-23der-miR-9aUCUUUGGUUAU-CUAGCUGUAUGA-23ssc-miR-9-2UCUUUGGUUAU-CUAGCUGUAUGA-23dvi-miR-9aUCUUUGGUUAU-CUAGCUGUAUGA-23ssc-miR-9-1UCUUUGGUUAU-CUAGCUGUAUGA-23dsi-miR-9aUCUUUGGUUAU-CUAGCUGUAUGA-23rno-miR-9-3UCUUUGGUUAU-CUAGCUGUAUGA-23dse-miR-9aUCUUUGGUUAU-CUAGCUGUAUGA-23rno-miR-9-2UCUUUGGUUAU-CUAGCUGUAUGA-23dme-miR-9aUCUUUGGUUAU-CUAGCUGUAUGA-23rno-miR-9-1UCUUUGGUUAU-CUAGCUGUAUGA-23dgr-miR-9aUCUUUGGUUAU-CUAGCUGUAUGA-23spu-miR-9UCUUUGGUUAU-CUAGCUGUAUG--22dpe-miR-9aUCUUUGGUUAU-CUAGCUGUAUGA-23bta-miR-9-2UCUUUGGUUAU-CUAGCUGUAUG--22dps-miR-9aUCUUUGGUUAU-CUAGCUGUAUGA-23bta-miR-9-1UCUUUGGUUAU-CUAGCUGUAUG--22dmo-miR-9aUCUUUGGUUAU-CUAGCUGUAUGA-23oan-miR-9-2UCUUUGGUUAU-CUAGCUGUAUG--22xtr-miR-9a-1UCUUUGGUUAU-CUAGCUGUAUGA-23oan-miR-9-1UCUUUGGUUAU-CUAGCUGUAUG--22xtr-miR-9bUCUUUGGUUAU-CUAGCUGUAUGA-23mmu-miR-9-3UCUUUGGUUAU-CUAGCUGUAUGA-23sme-miR-9aUCUUUGGUUUU-CUAGCUGUAUGAU24sko-miR-9UCUUUGGUUAU-CUAGCUGUAU---21sme-miR-9bUCUUUGGUUGU-UUAGCUAUAUGA-25mmu-miR-9-2UCUUUGGUUAU-CUAGCUGUAUGA-23ame-miR-9bGCUUUGGUAAU-CUAGCUUUAUGA-23mmu-miR-9-1UCUUUGGUUAU-CUAGCUGUAUGA-23bmo-miR-9bGCUUUGGUAAU-CUAGCUUUAUGA-23ppy-miR-9-3UCUUUGGUUAU-CUAGCUGUAUGA-23tca-miR-9bGCUUUGGUAAU-CUAGCUUUAUGA-23ppy-miR-9-2UCUUUGGUUAU-CUAGCUGUAUGA-23aae-miR-9UCUUUGGU-AUUCUAGCUGUAGA--22ppy-miR-9-1UCUUUGGUUAU-CUAGCUGUAUGA-23aga-miR-9cUCUUUGGU-AUUCUAGCUGUAGA--22ptr-miR-9-2UCUUUGGUUAU-CUAGCUGUAUGA-23cqu-miR-9UCUUUGGU-AUUCUAGCUGUAGA--22ptr-miR-9-1UCUUUGGUUAU-CUAGCUGUAUGA-23dan-miR-9cUCUUUGGU-AUUCUAGCUGUAGA--22hsa-miR-9-3UCUUUGGUUAU-CUAGCUGUAUGA-23der-miR-9cUCUUUGGU-AUUCUAGCUGUAGA--22hsa-miR-9-2UCUUUGGUUAU-CUAGCUGUAUGA-23dgr-miR-9cUCUUUGGU-AUUCUAGCUGUAGA--22hsa-miR-9-1UCUUUGGUUAU-CUAGCUGUAUGA-23dme-miR-9cUCUUUGGU-AUUCUAGCUGUAGA--22(待续)514·研究论文·miRNA名称成熟序列碱基数量miRNA名称成熟序列碱基数量NameofmiRNAMaturesequenceBaseNameofMaturesequenceBasenumebermiRNAnumeberggo-miR-9UCUUUGGUUAU-CUAGCUGUAUGA-23dmo-miR-9cUCUUUGGU-AUUCUAGCUGUAGA--22mne-miR-9UCUUUGGUUAU-CUAGCUGUAUGA-23dme-miR-9bUCUUUGGUGAUUUUAGCUGUAUG--23dpe-miR-9cUCUUUGGU-AUUCUAGCUGUAGA--22dmo-miR-9bUCUUUGGUGAUUUUAGCUGUAUG--23dps-miR-9cUCUUUGGU-AUUCUAGCUGUAGA--22dpe-miR-9bUCUUUGGUGAUUUUAGCUGUAUG--23dse-miR-9cUCUUUGGU-AUUCUAGCUGUAGA--22dps-miR-9bUCUUUGGUGAUUUUAGCUGUAUG--23dvi-miR-9cUCUUUGGU-AUUCUAGCUGUAGA--22dse-miR-9bUCUUUGGUGAUUUUAGCUGUAUG--23dwi-miR-9c-1UCUUUGGU-AUUCUAGCUGUAGA--22dvi-miR-9bUCUUUGGUGAUUUUAGCUGUAUG--23dwi-miR-9c-2UCUUUGGU-AUUCUAGCUGUAGA--22dwi-miR-9b-1UCUUUGGUGAUUUUAGCUGUAUG--23dya-miR-9cUCUUUGGU-AUUCUAGCUGUAGA--22dwi-miR-9b-2UCUUUGGUGAUUUUAGCUGUAUG--23bmo-miR-9cUCUUUGGU-AUCCUAGCUG------18dya-miR-9bUCUUUGGUGAUUUUAGCUGUAUG--23aae-miR-9bUCUUUGGUGAUUUUAGCUGUAUGC-23aga-miR-9bACUUUGGUGAUUUUAGCUGUAUG--23dan-miR-9bUCUUUGGUGAUUUUAGCUGUAUG--23dgr-miR-9bUCUUUGGUGAUUUUAGCUCUAUG--23der-miR-9bUCUUUGGUGAUUUUAGCUGUAUG--23Clustalconsensus阴影区域表示保守碱基,"-"表示碱基缺失.
Theshadowregionrepresenthighlyconservedbases,hyphen(-)indicatebasedelection.
串联重复后形成了紧密相连的mir-9b、mir-9c基因簇,随后经历片段缺失和碱基突变以mir-9a/9、mir-9b的形式存在于节肢动物门和线虫纲等无脊椎动物中.
在由无脊椎动物向脊椎动物进化的过程中mir-9/9a基因可能发生片段重复形成了低等脊椎动物(鱼纲)中的mir-9-1至mir-9-7基因.
2.
4靶基因的预测大量的研究表明,一些miRNA在转录后水平调控脑部基因的表达,而且在神经发育各个阶段都起着重要的调控作用.
miRNA在中枢神经系统的表达具短暂性和区域性[12].
如miR-138和miR-124主要在中枢神经系统中表达,miR-126和miR-29主要在星形胶质细胞中表达.
据报道miR-9是一个组织特异性的miRNA,它主要在中后脑交界处(midbrain-hindbrainboundary,MHB)的邻近区域表达[13],可通过作用于成纤维生长因子(fibroblastgrowthfactor,Fgf)信号通路调控MHB的组织活性,而且miR-9与神经发生、心脏肥大[14]、脑部正常发育[15,16]等具有密切关系.
为进一步了解miR-9参与的调控机制及miRNA作用的新标靶,本文以小鼠的mmu-miR-9基因为研究对象,采用TargetScan、PicTar、microrna.
org和MicroCosmTargrts等四个microRNA靶位点分析软件搜索mmu-miR-9的靶基因.
利用TheGeneOntol-ogy、KyotoEncyclopediaofGenesandGenomes和David软件分析四个靶基因软件均能预测到的靶基因(表3)的功能,从而了解miR-9在生物体中可能的作用路径.
由TheGeneOntology和KyotoEncyclopediaofGenesandGenomes数据库分析可知,miR-9的靶基因多数参与新陈代谢、信号转导、跨膜运输和物质转运等生命活动过程,与神经细胞、心肌细胞等细胞的生长和分化密切相关.
例如:nidogen2所编码的巢蛋白是神经干细胞的标志蛋白,Erbb2ip基因所编码的Erbb2相互作用蛋白是神经干细胞正常生长所必须的表皮生长因子受体(epidermalgrowthfactorreceptor,EGFR),Slc31a2所编码的铜离子转运蛋白对于维持神经系统的正常生理功能非常重要.
可见,miR-9在基因表达调控中具有广泛的作用.
Chun-nian等[17]曾报道miR-9对于神经干细胞的增殖和分化具有调控作用,Krichevsky等[18]在研究胚胎神经干细胞形成时发现以miR-9和miR-124为代表在脑部含量丰富的miRNA,主要是在神经细胞向神经元的分化过程中诱导产生的,而且在神经分化过程中阻断它们的功能会提升神经干细胞向星形胶质细胞分化的可能性.
这些研究工作也支持本文获得的结果.
David数据库的Functionannotationchart的数据统计显示mmu-miR-9的候选靶基因中,与磷酸化和乙酰化作用相关的基因数量最多,暗示着miR-9与表观调控路径相关,为我们研究mir-9的功能提供一个新的切入点.
(续表2)罗丹丹等:miRNA-9基因家族进化分析及其靶基因预测515图1MEGA4.
1软件NJ法构建mir-9基因家族系统进化树缩写含义:bfl:文昌鱼;xtr:爪蟾属;gga:原鸡;cfa:家犬;mdo:短尾猊;lla:绒毛猴属;mml:恒河猴;ggo:大猩猩;has:智人;ptr:黑猩猩属;oan:鸭嘴兽;mmu:小家鼠;bta:欧洲牛;ssc:野猪;dre:斑马鱼;spu:紫色球海胆;sko:Saccoglossuskowalevski;dpu:水蚤属;aga:冈比亚按蚊;dpe:果蝇;dvi:果蝇;lmi:飞蝗;tca:拟谷盗属;cap:Capitellasp.
I;lgi:Lottiagigantean;sme:Schmidteamediterranea.
Fig.
1phylogenetictreeofthemiR-9genefamilyconstructedbyMEGA4.
1softwarebasedonNeighbor-joiningmethodMeaningsofabbreviations:bfl,Branchiostomafloridae;xtr,Xenopustropicalis;gga,Gallusgallus;cfa,Canisfamiliaris;mdo,Monodelphisdomes-tica;lla,Lagothrixlagotricha;mml,Macacamulatta;ggo,Gorillagorilla;has,Homosapiens;ptr,Pantroglodyte;oan,Ornithorhynchusanatinus;mmu,Musmusculus;bta,BosTaurus;ssc,Susscrofa;dre,Daniorerio;spu,Strongylocentrotuspurpuratus;sko,Saccoglossuskowalevski;dpu,Daphniapulex;aga,Anophelesgambiae;dpe,Drosophilapseudoobscura;dvi,Drosophilavirilis;lmi,Locustamigratoria;tca,Triboliumcastaneum;cap,Capitellasp.
I;lgi,Lottiagigantean;sme,Schmidteamediterranea.
516·研究论文·表3四个靶基因分析软件均能预测到的mmu-miR-9靶基因Table3Targetsofmmu-miR-9predictedbyfoursoftwaresNCBI序列号基因名称编码蛋白所处代谢路径及其功能简介Acc.
No.
NameofgenesIntroductionofgenefunctionNM_025286Slc31a2,solutecarrierfamily31,member2Solutetransportfamilyof31,alsoknownascopperiontransportprotein,centralnervoussystemexistenceahighconcentrationofcopperion(Cu2+),andCu2+playaroleinthenerveterminalwhichcandepolarizedfromthesuddenreleasedintothesynapticvesiclescontactgapLaminA,themaincomponentofnuclearlamina,tomaintainlevelsinthenucleusplaysasupportingrole,asthenuclearmembraneandchromatinstructureprovidessupport.
NM_001002011Lmna,laminAMainlyinhypertrophiccardiomyopathy,arrhythmogenicrightventricularNM_011960Parg,poly(ADP-ribose)glycohydrolasecardiomyopathy,cardiomyopathyamplificationpathwaycatalyticactivity,Hydrolyzespoly(ADP-ribose)atglycosidic(1''-2')linkageofribose-ribosebondtoproducefreeADP-ribose.
,function,Poly(ADP-ribose)synthesizedafterDNAdamageisonlypresenttransientlyandisrapidlydegradedbypoly(ADP-riboseglycohydrolase.
Poly(ADP-ribose)metabolismmayberequiredformaintenanceofthenormalfunctionofneuronalcells.
,sequencecaution,TranslatedasMet.
,similarity,Belongstothepoly(ADP-ribose)glycohydrolasefamilyNM_146151Tesk2,testis-specifickinase2Testis-specificproteinkinase2,mainlycontainingN-terminalkinasedomainofserine/threonineproteinkinase,mainlyexpressedintestisandprostateNM_030706Trim2,tripartitemotifprotein2Tripartitemotifprotein2,expressionofihighlevelsinthenervoussystemtomaintainitsstructuralplasticityofneuronsNM_019767Arpc1a,actinrelatedprotein2/3complex,Actinrelatedprotein2/3complexandpromotemicrofilamentformationoflivingcellssurroundingMainlyinvolveinFCgammaR-relatedShijuneffect,adjusttheactincytoskeleton,bacterialmetabolicpathwayssuchasepithelialcellsinfectedNM_199029Zfp395,zincfingerprotein395ZincFingerprotein395,andiscloselyrelatedtoembryonicdevlopmentandcelldifferentiationisanimportantfunctionofaclassoftranscriptionfactorNM_031397Bicc1,bicaudalChomolog1(Drosophila)Two-tail-Cgenehomolog1,playaroleintheheadandtailstructure,theorgandevelopmentNM_011843Mbc2,membraneboundC2domaincontainingMembraneboundC2domaincontainingproteinproteinNM_008695Nid2,nidogen2Nidogen2,oneofembryonicintermediatefilamentprotein,whichisconsideredamarkerofneuralstemcellsNM_021563Erbb2ip,Erbb2interactingproteinErbb2interactingproteinsforepidermalgrowthfactorreceptor(epidermalgrowthfactorreceptor,EGFR)MainlyinvolveinNOD-likereceptorsprimarilysignalpathwayNM_016673Cntfr,ciliaryneurotrophicfactorreceptorCiliaryneurotrophicfactorreceptors,onnervecellgrowth,differentiationhasobviousnutritionalroleMainlyinvolveincytokinesandcytokinereceptorinteractionpathway,Jak-STATsignalingpathwayNM_023245Palmd,palmdelphinPalmdelphin,mainlyinthebrain,thehippocampus,amygdalaNM_134134A630042L21Rik,RIKENcDNAA630042L21geneHighmobilitygroupboxdomaincontainingfamilyofcomplex3NM_013890Fbxw2,F-boxandWD-40domainprotein2F-boxandWD-40domainprotein2NM_009679Ap2m1,adaptorproteincomplexAP-2,mu1AdaptorProteinComplexAP-2,μ1subunit,mainlycatalytic,transportprotein,proteinbinding,lipidbindingMainlyinvolveinendocytosis,Huntingtondiseaseandothermetabolicpathway(待续)罗丹丹等:miRNA-9基因家族进化分析及其靶基因预测517NCBI序列号基因名称编码蛋白所处代谢路径及其功能简介Acc.
No.
NameofgenesIntroductionofgenefunctionNM_134060Slc35b3,solutecarrierfamily35,memberB3Family35solutetransport,membranetransportproteinfamilymember,playsanimportantroleinneuralstemcellrenewalandneurogenesisNM_026139Armcx2,armadillorepeatcontaining,X-linked2Armadillorepeatcontaining,X-linked2,astrongsignalinembryonicdevelopmentstageNM_011945Map3k1,mitogenactivatedproteinkinaseMitogen-activatedproteinkinasekinase1,throughthekinasekinase1MAPKcascadecontrolledresponsesysteminvolvedincellproliferationanddifferentiation,whenthevitalityoutofcontrolwillleadtotumorMainlyinvolveinMAPKsignalpathway,ubiquitin-mediatedproteindegradation,RIG-I-likereceptorsignalingpathway,neurotrophicfactorsignalingpathway,GnRHsignalingpathwayandotherrelatedNM_015736Galnt3,UDP-N-acetyl-alpha-D-galactosamineN-acetylgalactoseaminotransferasepolypeptide,oftenwiththefibroblastgrowthfactor23witheffectMainlyinvolveinO-polysaccharidebiosynthesis,metabolicpathwayNM_009680Ap3b1,adaptor-relatedproteincomplex3,Adaptor-relatedproteincomplex3,beta1subunit,ismainlycatalytic,beta1subunittransportprotein,proteinbinding,lipidbindingMajormetabolicpathwaysassociatedwiththelysosomalNM_146164Lrch4,leucine-richrepeatsandcalponinhomologyLeucine-richrepeatsandcalponinhomology(CH)domaincontaining4,(CH)domaincontaining4embryonicgrowthanddifferentiationofspecificreceptorNM_146087Csnk1a1,caseinkinase1,alpha1Caseinkinase1,intheWntpathwayplaysanimportantroleinMainlyinvolveinWntsignalingpathway,HedgehogsignalingpathwayNM_023670Igf2bp3,insulin-likegrowthfactor2,bindingInsulin-likegrowthfactor2,bindingprotein3protein3NM_1786134933433P14Rik,UPF0279proteinRIKENcDNNA4933433P14geneC14orf129homolog.
NM_133195Brunol4,bruno-like4,RNAbindingproteinBruno-like-Gene4,RNAbindingprotein,playaroleinalternativesplicingandthetargetmRNAtranslationandstability3讨论microRNA作为重要的内源性调控基因,分布范围广,参与的生物学过程复杂,调控靶基因数众多,对于生物的正常生长发育至关重要,在动植物的进化过程中扮演着重要的角色.
mir-9基因家族在原口动物和后口动物中分布广泛,且高度保守,说明mir-9基因在生物起源和迁移过程中的重要作用.
mir-9基因家族成熟序列的2-8位碱基即"种子区域"的保守性为100%,暗示其功能的保守性.
从低等向高等进化过程中普遍存在mir-9基因家族的调控机制,说明miRNA的调控机制对物种生存的重要性[19].
Maller等[20]在通过分析拟南芥基因组,发现许多miRNAs家族是通过基因组水平上的复制、串联重复或者大片段复制产生的,接着再发生时空表达的分化.
Tanzer等[21]发现miR-17基因簇进化历史伴随着串联重复、整个基因簇复制以及个别miRNA的随机缺失,而且miR-17基因家族可能与脊椎动物的早期进化有关.
对于mir-9基因的进化分析显示,mir-9、mir-9b、mir-9c、mir-9a基因主要出现在进化早期,为祖先基因,且mir-9b和mir-9c聚集成簇.
Hertel等[22]在后生动物基因组中的miRNA基因家族之间的系统发育分析揭示了串联重复和片段重复是miRNA基因簇进化的主要机制,也因此产生了miRNA基因簇和miRNA基因家族的多样性分布.
由此推测miR-9b和miR-9c两个基因在同一染色体上所形成的基因簇可能是由串联重复所产生的.
mir-9-1至mir-9-7则主要分布在两栖类及哺乳类等较高等的脊椎动物中,mir-9基因家族从无脊椎动物向脊椎动物进化的过程中数量有所增多,可能发生了串联重复、大片段重复和个别miRNA的缺失.
所以对mir-9的系统进化分析不仅有利于揭示非编码小分子RNA的起源及其作用机制,还可以增进对物种进(续表3)518·研究论文·化的了解.
Kosik等[23]和Landgraf等[24]对于microRNA的研究显示,miRNA在神经系统中含量丰富,表达具有组织特异性和非对称性,说明miRNAs在神经系统的特定功能中所扮演的重要角色.
Monika等[5]利用原位杂交技术发现miR-124及miR-9在神经系统中表达丰富,miR-9主要在神经前体细胞和神经元中表达,它是神经发育和成脑中所出现的特异性miRNA[25,26].
本文利用生物信息学方法,在四个miRNA在线分析软件中搜索miR-9的共同靶基因,通过GO和KEGG分析显示mir-9基因家族可通过其靶基因作用于多条代谢路径,且与神经系统正常发育与生长途径具有密切关系.
所以对于mir-9基因家族的靶基因的预测不仅有利于了解其可能参与的生物学途径,还可为以后的实验研究提供理论依据.
参考文献(References)1DennisC.
ThebravenewworldofRNA.
Nature2002;418(6894):122-4.
2RuvkunG.
Molecularbiology,glimpsesofatinyRNAworld.
Science2001;294(5543):797-9.
3LauNC,LimLP,WeinsteinEG,BartelDP.
AnabundantclassoftinyRNAswithprobableregulatoryrolesinCaenorhabditiselegans.
Science2001;294(5543):858-62.
4RodriguezA,Griffiths-JonesS,AshurstJL,BradleyA.
Identi-ficationofmammalianmicroRNAhostgenesandtranscriptionunits.
GenomeRes2004;14(10A):1902-10.
5DeoM,YuJY,ChungKH,TippensM,TurnerDL.
DetectionofmammalianmicroRNAexpressionbyinsituhybridizationwithRNAoligonucleotides.
DevDyn2006;235(9):2538-48.
6YooAS,StaahlBT,ChenL,CrabtreeGR.
MicroRNA-mediatedswitchingofchromatin-remodellingcomplexesinneuraldevel-opment.
Nature2009;460(7255):642-6.
7DelaloyC,LiuL,LeeJA,SuH,ShenF,YangGY,etal.
Mi-croRNA-9coordinatesproliferationandmigrationofhumanembryonicstemcell-derivedneuralprogenitors.
CellStemCell2010;6(4):323-35.
8MaL,YoungJ,PrabhalaH,PanE,MestdaghP,MuthD,etal.
miR-9,aMYC/MYCN-activatedmicroRNA,regulatesE-cadher-inandcancermetastasis.
NatCellBiol2010;12(3):247-56.
9TavazoieSF,AlarcónC,OskarssonT,PaduaD,WangQ,BosPD,etal.
EndogenoushumanmicroRNAsthatsuppressbreastcancermetastasis.
Nature2008;451(7175):147-52.
10DoenchJG,SharpPA.
SpecificityofmicroRNAtargetselectionintranslationalrepression.
GenesDev2004;18(5):504-11.
11SaitouN,NeiM.
Theneighbor-joiningmethod:anewmethodforreconstructingphylogenetictrees.
MolBiolEvol1987;4:406-25.
12WangJF,ZhouH,ChenYQ,LuoQJ,QuLH.
Identificationof20microRNAsfromOryzasativa.
NucleicAcidsRes2004;32(5):1688-95.
13DelaloyC,GaoFB.
microRNA-9multitaskingnearorganizingcenters.
NatNeurosci2008;11:625-6.
14WangK,LongB,ZhouJ,LiPF.
miR-9andNFATc3regulatemyocardinincardiachypertrophy.
JBiolChem2010;285(16):11903-12.
15ZhangZ,ChangH,LiY,ZhangT,ZouJ,ZhengX,etal.
Mi-croRNAs:potentialregulatorsinvolvedinhumananencephaly.
IntJBiochemCellBiol2010;42(2):367-74.
16CoolenM,Bally-CuifL.
MicroRNAsinbraindevelopmentandphysiology.
CurrOpinNeurobiol2009;19(5):461-70.
17ZhaoC,SunG,LiS,ShiY.
AfeedbackregulatoryloopinvolvingmicroRNA-9andnuclearreceptorTLXinneuralstemcellfatedetermination.
NatStructMolBiol2009;16(4):365-71.
18KrichevskyAM,SonntagKC,IsacsonO,KosikKS.
SpecificmicroRNAsmodulateembryonicstemcell-derivedneurogenesis.
StemCells2006;24:857-64.
19MassirerKB,PasquinelliAE.
TheevolvingroleofmicroRNAsinanimalgeneexpression.
Bioessays2006;28:449-52.
20MaherC,SteinL,WareD.
EvolutionofArabidopsismicroRNAfamiliesthroughduplicationevents.
GenomeRes2006;16:510-9.
21TanzerA,StadlerPF.
MolecularevolutionofamicroRNAclus-ter.
JMolBiol2004;339:327-35.
22HertelJ,LindemeyerM,MissalK,FriedC,TanzerA,FlammC,etal.
TheexpansionofthemetazoanmicroRNArepertoire.
BMCGenomics2006;7:25.
23KosikKS.
TheneuronalmicroRNAsystem.
NatRevNeurosci2006;7(12):911-20.
24LandgrafP,RusuM,SheridanR,SewerA,IovinoN,AravinA,etal.
AmammalianmicroRNAexpressionatlasbasedonsmallRNAlibrarysequencing.
Cell2007;129:1401-14.
25Lagos-QuintanaM,RauhutR,YalcinA,MeyerJ,LendeckelW,TuschlT.
Identificationoftissue-specificmicroRNAsfrommouse.
CurrBiol2002;12:735-9.
26KrichevskyAM,KingKS,DonahueCP,KhrapkoK,KosikKS.
AmicroRNAarrayrevealsextensiveregulationofmicroRNAsduringbraindevelopment.
RNA2003;9(10):1274-81.
罗丹丹等:miRNA-9基因家族进化分析及其靶基因预测519MolecularEvolutionofmiRNA-9GeneFamilyandPredictionofTheirTargetGenesDan-DanLuo,Yu-PingLuo,JuanPeng,Si-GuangLi*(CollegeofLifeSciencesandFoodEngineering,NanchangUniversity,Nanchang330031,China)AbstractmicroRNAsareaclassofnon-codingsmallRNAsof21~25ntsineucaryota,whichregulategeneexpressionatthepost-transcriptionallevel.
Inthisstudy,wesearchedmir-9genesofmetazoainmiRBase,andgotatotalof120sequencesin47specieswhichindicatingtheextensiveexistenceofmir-9geneindifferentspecies.
Theanalysisofgenelocalizationshown86%ofmir-9geneslocateintheintergenicregion(IGR).
Multiplesequencealignmentofmaturesequencesofmir-9genefamilyshowedthatthesecondtoeighthbasesandthefour-teenthtoeighteenthbasesareconservativebases.
Phylogeneticanalysisrevealedmir-9bandmir-9cmaybetheear-liestgeneformsofthisfamily,viz.
ancestralgenesofmir-9family.
Theseancestralgenescreatedmir-9-1tomir-9-7genesinvertebratebytandemduplication,largersegmentduplication,deletionandmutationofindividualbases.
Targetgenesofmmu-miR-9arepredictedbyfourmiRNAtargetgenepredictionsoftwares.
TheresultshowedthatmiR-9wasinvolvedinnervoussystemdevelopment,cardiacdiseasesandtransmembranetransportsystem.
TheseresultscontributetofurtherstudyontheroleofmicroRNAinnervoussystemdevelopmentandthemechanismofneuralcellgrowthanddifferentiation.
KeywordsmiRNA-9;evolution;targetgeneReceived:October11,2011Accepted:February25,2011ThisworkwassupportedbytheNationalNaturalScienceFoundationofChina(No.
30971473,No.
31071138)*Correspondingauthor.
Tel:86-791-8304099,E-mail:siguangli@163.
com
cjcb.
orgmiRNA-9基因家族进化分析及其靶基因预测罗丹丹罗玉萍彭娟李思光*(南昌大学生命科学与食品工程学院,南昌330031)摘要microRNAs(miRNA)是真核生物中一类长度约为21~25个核苷酸的非编码小分子RNA,在转录后水平调控基因的表达.
该文在miRBase中搜索后生动物的mir-9基因序列.
47个物种中共搜索到120条mir-9基因序列,说明mir-9基因家族广泛存在于不同物种中.
基因定位显示86%的mir-9基因存在于基因间隔区(IGR),多序列比对发现miR-9基因家族成熟序列的第2位到第8位碱基以及第14位到第18位碱基为保守碱基.
进化分析表明mir-9b和mir-9c可能是此基因家族最早出现的基因形式,即祖先基因.
这些祖先基因经过串联重复、大片段重复、个别碱基的缺失及突变等方式形成了脊椎动物中miR-9-1至miR-9-7数个基因.
分别采用四个miRNA靶基因预测软件对mmu-miR-9的靶基因进行预测,发现miR-9与神经系统发育、心肌系统疾病和跨膜运输系统等密切相关.
该研究为今后进一步研究miRNA调控的神经系统发生和神经细胞生长与分化的机制奠定了基础.
关键词miRNA-9;进化;靶基因收稿日期:2010-10-11接受日期:2011-02-25国家自然科学基金(No.
30971473,No.
31071138)资助项目*通讯作者.
Tel:0791-8304099,E-mail:siguangli@163.
commiRNAs是一类长度为20~28nt的非编码小分子RNA,通过与靶mRNA的互补配对而在转录后水平上负调控基因的表达[1].
miRNA基因以单拷贝、多拷贝和基因簇等多种形式存在于基因组中.
成簇基因之间有的具有同源性,有的无同源性,其中串联基因簇(tandemcluster)中的miRNA一般呈现为协同表达[2,3],暗示着miRNA基因存在复杂的调控机制.
早期的研究认为miRNA基因主要存在于基因间隔区,但近期的研究发现大部分哺乳动物的miRNA基因主要定位在转录单位(transcriptunits,TUs)[2,4],且大多数位于内含子区.
不同物种间的同源miRNA存在高度进化保守性.
如miR-1、miR-34、miR-87在无脊椎动物和脊椎动物中高度保守,序列比对分析发现这些保守序列的碱基差异仅为1-2nt.
Lau等[3]在美丽新小杆线虫(Caenorhabditiselegans)中发现的miRNA,85%在C.
briggsaze基因组序列中具有同源序列.
miRNA在物种间的保守性使其成为探索非编码RNA进化规律的理想分子.
已有研究报道,小鼠和人等哺乳动物中miR-9主要在神经组织中表达[5],并通过调控BAF53a[6]、stathmin[7]等基因的表达参与神经细胞的增殖、迁移等活动.
近期研究显示miR-9还控制着乳腺肿瘤的扩散和发展[8,9],所以,对miR-9的深入研究不仅利于揭示其所介导的调控网络,还将为人类疾病的诊断和治疗提供全新的视野.
本文根据miRNA的进化保守性,通过多序列比对及构建系统进化树的方式分析mir-9基因家族的进化历史.
采用TargetScan、PicTar、microRNA.
org和MicroCosmTargrts等四个miRNA靶基因在线分析软件分析miR-9的靶基因.
通过TheGeneOntology、KyotoEncyclopediaofGenesandGenomes和David软件分析了靶基因的生物学功能,进一步了解miR-9在生物体内的角色及其可能的作用机制.
1材料与方法1.
1mir-9序列的获得从miRBase(http://www.
mirbase.
org/cgi-bin/browse.
pl)中下载部分后生动物的mir-9基因前体序列及成熟序列.
1.
2几种模式生物的mir-9基因定位采用NCBI数据库BLAST(http://blast.
ncbi.
nlm.
罗丹丹等:miRNA-9基因家族进化分析及其靶基因预测511nih.
gov/Blast.
cgi)程序,分别用人、黑猩猩、牛、小鼠、斑马鱼、原鸡和黑腹果蝇等7个模式物种的miRNA前体序列对其基因组数据库进行搜索(BLASTAs-sembledRefSeqGenomes),以确定miR-9在基因组中的位置.
1.
3系统发生分析用ClustalX1.
83软件对所有已搜索物种的mir-9基因的前体序列进行多序列比对分析(multiplese-quencealigment,MSA).
利用MEGA4.
0软件采用基于距离参数的邻接法(Neighbor-joining,NJ),并自展分析(Bootstrap)1000次,构建各个纲目中模式生物的系统进化树.
1.
4靶基因的预测以mmu-miR-9(小鼠miR-9)为研究对象,通过TargetScan(http://www.
targetscan.
org/mmu_50/)、PicTar(http://pictar.
mdc-berlin.
de/cgi-bin/new_PicTar_mouse.
cgispecies=mouse)、microrna.
org(http://www.
ebi.
ac.
uk/enright-srv/microcosm/cgi-bin/targets/v5/search.
pl)及MicroCosmTargrts(http://www.
ebi.
ac.
uk/enright-srv/microcosm/cgi-bin/targets/v5/search.
pl)四个microRNA靶位点分析软件在线分析mmu-miR-9的靶基因,选取四个软件均能预测到的靶基因作为候选靶基因.
通过分析靶基因的功能进一步认识mmu-miR-9的生物学功能.
1.
5靶基因的功能分析利用TheGeneOntology(http://www.
geneontology.
org/)、KyotoEncyclopediaofGenesandGenomes(http://www.
genome.
jp/kegg/)和David(http://david.
abcc.
ncifcrf.
gov/)数据库分析靶基因的生物学功能.
2结果2.
1miR-9基因家族及其在基因组中的定位在miRBase中搜索后生动物的mir-9基因序列,在47个物种中共搜索到120条mir-9基因同源序列.
其中,斑马鱼(Daniorerio)的序列数量最多,共7条,原鸡(Gallusgallus)、大猩猩(Gorillagorilla)、飞蝗(Locustamigratoria)等只有一条或者两条序列.
值得注意的是,人类mir-9在miRBase中存在has-mir-9-1、has-mir-9-2和has-mir-9-3三条序列,而同为人类近亲的大猩猩(Gorillagorilla)和黑猩猩(Pantroglodytes)在miRBase数据库中分别只有一条和两条mir-9序列.
利用has-mir-9的前体序列作为查询序列,采用BLAST程序分别对黑猩猩和大猩猩基因组数据库进行搜索,发现在黑猩猩(Pantroglodytes)中还存在一条miRBase中没有收录的has-mir-9-3的同源序列,在大猩猩(Gorillagorilla)中还存在一条miR-Base没有收录的hsa-mir-9-2的同源序列,mfold折叠证实这些序列都存在miRNA前体典型的二级结构,说明这些物种中还存在目前尚未发现的新的mir-9基因.
以人、黑猩猩、牛、小鼠、斑马鱼、原鸡和黑腹果蝇等7个模式生物mir-9前体序列作为查询序列(QuerySequence),分别对这些生物的基因组DNA进行BLAST搜索,确定各序列在基因组中的位置(表1).
基因定位显示,与其它哺乳动物的miRNA主要定位在内含子区域的情况不同,86%的mir-9基因家族成员位于基因间隔区(intergenicregion,IGR),少数位于蛋白编码基因的内含子区域;mir-9b、mir-9c两个基因位于同一染色体上,并与mir-306、mir-79紧密相连聚集成簇;mir-9a、mir-9b和mir-9c主要出现在较低等的模式生物黑腹果蝇中,而mir-9-1至mir-9-7则主要存在于哺乳类、鸟类、鱼类等脊椎动物中.
mir-9a、mir-9b和mir-9c主要出现在物种进化的早期或低等生物中的现象表明其可能为mir-9家族中较原始的基因.
2.
2miR-9基因家族序列分析将mir-9基因家族的成熟序列进行多序列比对分析(表2),发现为第2位到第8位碱基以及第14位到第18位碱基为保守碱基,说明miR-9基因在不同物种间的高度保守性.
比对结果显示miR-9在其进化过程中可能发生了缺失:有的物种在第9位缺失了碱基A/U/G,有的物种在第23位缺失了碱基A;大多数物种在第12位缺失了碱基U,第24位缺失了碱基U.
第9~11位碱基的相对不保守性可能是因为三个碱基的错配结构更有利于其与靶序列的配对[10].
序列比对发现miR-9与miR-9a的成熟序列相同或只在3'末端相差一个碱基A,但在所有物种的前体序列的相应位置都具有碱基A,所以miR-9与miR-9a可能是相同的miRNA,只是由于3'端的A容易脱落,不同研究者在实验中得到了3'端相差1个A的两种分子.
作为转录后水平调控元件,miRNA成熟序列5'端的第2-8位碱基对于其功能的正常发挥至关重要,被称为"种子序列(seedsequence)"[10].
种子序列主要通过与目标mRNA3'UTR区域的互补配对识别512·研究论文·靶基因,负调控靶基因的表达.
在同源性miRNA中,这段序列通常是保守的,我们的比对结果也证实了这点.
mir-9家族前体序列的同源性分析表明,人与黑猩猩的同源性为100%,人与恒河猴之间的同源性为85%,而人与较低等的两栖类(非洲爪蟾)的同源性约为58%,与果蝇的同源性只有39%.
可见亲缘关系近的物种之间核苷酸序列差异小,亲缘关系远的物种之间核苷酸序列差异大.
仔细分析miR-9的前体序列发现,序列差异主要发生在miR-9前体中成熟序列以外的区域,miRNA成熟序列的区域由于受到选择压力而高度保守.
2.
3miR-9基因家族系统进化树的构建来源于miRBase中的miR-9基因主要分布在哺乳类、鸟类、鱼类、两栖纲和节肢动物门、线虫纲中.
采用MEGA4.
0软件,以这些纲目中的模式物种的mir-9序列构建基于距离参数的邻接法(Neighbor-joining,NJ)[11]构建系统发育进化树(图1).
图1可见,进化树可分为三支:mir-9、mir-9b、mir-9c主要分布在紫色球海胆、Capitellasp.
I(线虫纲)、果蝇(节肢动物门)等无脊椎低等生物中,聚为第一支.
从进化树的分支和距离来看,mir-9、mir-9b、mir-9c出现在进化早期并持续到现在,由此可以推测mir-9、mir-9b、mir-9c可能是mir-9基因家族最早出现的基因形式,即为祖先基因.
而且mir-9b和mir-9c位于同一分支中,说明它们亲缘关系近.
基因定位分析显示mir-9b和mir-9c聚集成簇,暗示它们在进化过程中紧密相连.
第二支主要是分布在节肢动物门的mir-9a及部分环节动物门和线虫门的mir-9和mir-9b.
第三分支主要为人、黑猩猩、鱼类、鸟类等脊椎动物的mir-9基因家族成员.
在这一支中,除同源性较低、亲缘关系较远的文昌鱼(全索亚门)的mir-9单列一小支,其他物种中的mir-9-2、mir-9-5、mir-9-6属同一亚支,而mir-9-1、mir-9-3、mir-9-4、mir-9-7属与之平行进化的另一亚支.
分析发现,miR-9b和miR-9c两个拷贝主要出现在无脊椎动物中,miR-9-1、miR-9-2、miR-9-3、miR-9-4、miR-9-5、miR-9-6和miR-9-7主要出现在脊椎动物中,miR-9和miR-9a在脊椎和无脊椎动物中都有分布.
根据上述的分析和比对结果,可以推测mir-9c作为该基因家族最原始的基因存在于线虫纲,经表1miR-9基因家族在七个物种中的基因位置示意图Table1GenomiclocationofmiRNA-9genefamilyinsevenspecies物种miRNA基因名称染色体基因位置SpeciesNameofmiRNAChromosomeGenepositionHomosapienshas-mir-9-1ChrIIntronofClorf61genehas-mir-9-2chr5IGRbetweenMEF2CandTMEM161Bgenehas-mir-9-3chr15IGRbetweenLoc100288642andLoc254559genePantroglodytesptr-mir-9-1chr1IntronofClorf61geneptr-mir-9-2chr5IGRbetweenMEF2CandTMEM161BgenePtr-mir-9-3chr15IGRbetweenPLOGandLOC453637geneMousemmu-mir-9-1chr3IntronofLoc10042277mmu-mir-9-2chr13IGRbetweenMEF2CandLoc62324genemmu-mir-9-3chr7IGRbetweenPlogandRhcggeneGallusgallusgga-mir-9-1chr21IGRbetweenLOC420111andLOC427091genegga-mir-9-2chrZIGRbetweenLOC769007andLOC426919geneBostaurusbta-mir-9-1chr21IGRbetweenLoc533090andRHCGgenebta-mir-9-2chr7IGRbetweenTMEM1618andMEF2cgeneDanioreriodre-mir-9-1chr16IGRbetweenRhb9andMef2dgenedre-mir-9-2chr10IGRbetweenLOC564822andMef2cagenedre-mir-9-3chr25IGRbetweenLOC560613andRhc9genedre-mir-9-4chr22IGRbetweentmem161aandgata2agenedre-mir-9-5chr5IGRbetweenmef2cbandtmem161bgenedre-mir-9-6chr7IGRbetweenZgc162132andKif7genedre-mir-9-7NOTFoundDrosophilamelanogasterdre-mir-9achr3LIGRbetweenCG93COandSha1genedre-mir-9b/9cchr21IntronofGrpgene,anddre-mir-9b/9cclusteringtogetherwithmir-306、mir-306s、mir-79罗丹丹等:miRNA-9基因家族进化分析及其靶基因预测513表2mir-9基因家族各成熟序列比对Table2Multiplesequencealigmentofmaturesequenceofmir-9genefamilymiRNA名称成熟序列碱基数量miRNA名称成熟序列碱基数量NameofmiRNAMaturesequenceBaseNameofMaturesequenceBasenumebermiRNAnumeberxtr-miR-9-3UCUUUGGUUAU-CUAGCUGUAUGA-22mml-miR-9-3UCUUUGGUUAU-CUAGCUGUAUGA-23ame-miR-9aUCUUUGGUUAU-CUAGCUGUAUGA-23mml-miR-9-2UCUUUGGUUAU-CUAGCUGUAUGA-23bfl-miR-9-2UCUUUGGUUAU-CUAGCUGUAUGA-22mml-miR-9-1UCUUUGGUUAU-CUAGCUGUAUGA-23bfl-miR-9-1UCUUUGGUUAU-CUAGCUGUAUGA-22lla-miR-9UCUUUGGUUAU-CUAGCUGUAUGA-23bmo-miR-9UCUUUGGUUAU-CUAGCUGUAUGA-23age-miR-9UCUUUGGUUAU-CUAGCUGUAUGA-23lgi-miR-9UCUUUGGUUAU-CUAGCUGUAUGA-23mdo-miR-9-2UCUUUGGUUAU-CUAGCUGUAUGA-23cap-miR-9UCUUUGGUUAU-CUAGCUGUAUGA-23mdo-miR-9-1UCUUUGGUUAU-CUAGCUGUAUGA-23tca-miR-9aUCUUUGGUUAU-CUAGCUGUAUGA-23eca-miR-9a-2UCUUUGGUUAU-CUAGCUGUAUGA-23lmi-miR-9aUCUUUGGUUAU-CUAGCUGUAUGA-23eca-miR-9aUCUUUGGUUAU-CUAGCUGUAUGA-23dpu-miR-9UCUUUGGUUAU-CUAGCUGUAUGA-23cfa-miR-9-3UCUUUGGUUAU-CUAGCUGUAUGA-23tni-miR-9-4UCUUUGGUUAU-CUAGCUGUAUGA-23cfa-miR-9-2UCUUUGGUUAU-CUAGCUGUAUGA-23tni-miR-9-3UCUUUGGUUAU-CUAGCUGUAUGA-23cfa-miR-9-1UCUUUGGUUAU-CUAGCUGUAUGA-23tni-miR-9-2UCUUUGGUUAU-CUAGCUGUAUGA-23gga-miR-9-2UCUUUGGUUAU-CUAGCUGUAUGA-23tni-miR-9-1UCUUUGGUUAU-CUAGCUGUAUGA-23gga-miR-9-1UCUUUGGUUAU-CUAGCUGUAUGA-23fru-miR-9-4UCUUUGGUUAU-CUAGCUGUAUGA-23tgu-miR-9-2UCUUUGGUUAU-CUAGCUGUAUGA-23fru-miR-9-3UCUUUGGUUAU-CUAGCUGUAUGA-23tgu-miR-9-1UCUUUGGUUAU-CUAGCUGUAUGA-23fru-miR-9-2UCUUUGGUUAU-CUAGCUGUAUGA-23xtr-miR-9a-2UCUUUGGUUAU-CUAGCUGUAUGA-23fru-miR-9-1UCUUUGGUUAU-CUAGCUGUAUGA-23dya-miR-9a-2UCUUUGGUUAU-CUAGCUGUAUGA-23dre-miR-9-7UCUUUGGUUAU-CUAGCUGUAUGA--23dya-miR-9a-1UCUUUGGUUAU-CUAGCUGUAUGA-23dre-miR-9-6UCUUUGGUUAU-CUAGCUGUAUGA-23aga-miR-9aUCUUUGGUUAU-CUAGCUGUAUGA-23dre-miR-9-5UCUUUGGUUAU-CUAGCUGUAUGA-23dan-miR-9aUCUUUGGUUAU-CUAGCUGUAUGA-23dre-miR-9-4UCUUUGGUUAU-CUAGCUGUAUGA-23dwi-miR-9aUCUUUGGUUAU-CUAGCUGUAUGA-23dre-miR-9-3UCUUUGGUUAU-CUAGCUGUAUGA-23aae-miR-9a-2UCUUUGGUUAU-CUAGCUGUAUGA-23dre-miR-9-2UCUUUGGUUAU-CUAGCUGUAUGA-23aae-miR-9a-1UCUUUGGUUAU-CUAGCUGUAUGA-23dre-miR-9-1UCUUUGGUUAU-CUAGCUGUAUGA-23der-miR-9aUCUUUGGUUAU-CUAGCUGUAUGA-23ssc-miR-9-2UCUUUGGUUAU-CUAGCUGUAUGA-23dvi-miR-9aUCUUUGGUUAU-CUAGCUGUAUGA-23ssc-miR-9-1UCUUUGGUUAU-CUAGCUGUAUGA-23dsi-miR-9aUCUUUGGUUAU-CUAGCUGUAUGA-23rno-miR-9-3UCUUUGGUUAU-CUAGCUGUAUGA-23dse-miR-9aUCUUUGGUUAU-CUAGCUGUAUGA-23rno-miR-9-2UCUUUGGUUAU-CUAGCUGUAUGA-23dme-miR-9aUCUUUGGUUAU-CUAGCUGUAUGA-23rno-miR-9-1UCUUUGGUUAU-CUAGCUGUAUGA-23dgr-miR-9aUCUUUGGUUAU-CUAGCUGUAUGA-23spu-miR-9UCUUUGGUUAU-CUAGCUGUAUG--22dpe-miR-9aUCUUUGGUUAU-CUAGCUGUAUGA-23bta-miR-9-2UCUUUGGUUAU-CUAGCUGUAUG--22dps-miR-9aUCUUUGGUUAU-CUAGCUGUAUGA-23bta-miR-9-1UCUUUGGUUAU-CUAGCUGUAUG--22dmo-miR-9aUCUUUGGUUAU-CUAGCUGUAUGA-23oan-miR-9-2UCUUUGGUUAU-CUAGCUGUAUG--22xtr-miR-9a-1UCUUUGGUUAU-CUAGCUGUAUGA-23oan-miR-9-1UCUUUGGUUAU-CUAGCUGUAUG--22xtr-miR-9bUCUUUGGUUAU-CUAGCUGUAUGA-23mmu-miR-9-3UCUUUGGUUAU-CUAGCUGUAUGA-23sme-miR-9aUCUUUGGUUUU-CUAGCUGUAUGAU24sko-miR-9UCUUUGGUUAU-CUAGCUGUAU---21sme-miR-9bUCUUUGGUUGU-UUAGCUAUAUGA-25mmu-miR-9-2UCUUUGGUUAU-CUAGCUGUAUGA-23ame-miR-9bGCUUUGGUAAU-CUAGCUUUAUGA-23mmu-miR-9-1UCUUUGGUUAU-CUAGCUGUAUGA-23bmo-miR-9bGCUUUGGUAAU-CUAGCUUUAUGA-23ppy-miR-9-3UCUUUGGUUAU-CUAGCUGUAUGA-23tca-miR-9bGCUUUGGUAAU-CUAGCUUUAUGA-23ppy-miR-9-2UCUUUGGUUAU-CUAGCUGUAUGA-23aae-miR-9UCUUUGGU-AUUCUAGCUGUAGA--22ppy-miR-9-1UCUUUGGUUAU-CUAGCUGUAUGA-23aga-miR-9cUCUUUGGU-AUUCUAGCUGUAGA--22ptr-miR-9-2UCUUUGGUUAU-CUAGCUGUAUGA-23cqu-miR-9UCUUUGGU-AUUCUAGCUGUAGA--22ptr-miR-9-1UCUUUGGUUAU-CUAGCUGUAUGA-23dan-miR-9cUCUUUGGU-AUUCUAGCUGUAGA--22hsa-miR-9-3UCUUUGGUUAU-CUAGCUGUAUGA-23der-miR-9cUCUUUGGU-AUUCUAGCUGUAGA--22hsa-miR-9-2UCUUUGGUUAU-CUAGCUGUAUGA-23dgr-miR-9cUCUUUGGU-AUUCUAGCUGUAGA--22hsa-miR-9-1UCUUUGGUUAU-CUAGCUGUAUGA-23dme-miR-9cUCUUUGGU-AUUCUAGCUGUAGA--22(待续)514·研究论文·miRNA名称成熟序列碱基数量miRNA名称成熟序列碱基数量NameofmiRNAMaturesequenceBaseNameofMaturesequenceBasenumebermiRNAnumeberggo-miR-9UCUUUGGUUAU-CUAGCUGUAUGA-23dmo-miR-9cUCUUUGGU-AUUCUAGCUGUAGA--22mne-miR-9UCUUUGGUUAU-CUAGCUGUAUGA-23dme-miR-9bUCUUUGGUGAUUUUAGCUGUAUG--23dpe-miR-9cUCUUUGGU-AUUCUAGCUGUAGA--22dmo-miR-9bUCUUUGGUGAUUUUAGCUGUAUG--23dps-miR-9cUCUUUGGU-AUUCUAGCUGUAGA--22dpe-miR-9bUCUUUGGUGAUUUUAGCUGUAUG--23dse-miR-9cUCUUUGGU-AUUCUAGCUGUAGA--22dps-miR-9bUCUUUGGUGAUUUUAGCUGUAUG--23dvi-miR-9cUCUUUGGU-AUUCUAGCUGUAGA--22dse-miR-9bUCUUUGGUGAUUUUAGCUGUAUG--23dwi-miR-9c-1UCUUUGGU-AUUCUAGCUGUAGA--22dvi-miR-9bUCUUUGGUGAUUUUAGCUGUAUG--23dwi-miR-9c-2UCUUUGGU-AUUCUAGCUGUAGA--22dwi-miR-9b-1UCUUUGGUGAUUUUAGCUGUAUG--23dya-miR-9cUCUUUGGU-AUUCUAGCUGUAGA--22dwi-miR-9b-2UCUUUGGUGAUUUUAGCUGUAUG--23bmo-miR-9cUCUUUGGU-AUCCUAGCUG------18dya-miR-9bUCUUUGGUGAUUUUAGCUGUAUG--23aae-miR-9bUCUUUGGUGAUUUUAGCUGUAUGC-23aga-miR-9bACUUUGGUGAUUUUAGCUGUAUG--23dan-miR-9bUCUUUGGUGAUUUUAGCUGUAUG--23dgr-miR-9bUCUUUGGUGAUUUUAGCUCUAUG--23der-miR-9bUCUUUGGUGAUUUUAGCUGUAUG--23Clustalconsensus阴影区域表示保守碱基,"-"表示碱基缺失.
Theshadowregionrepresenthighlyconservedbases,hyphen(-)indicatebasedelection.
串联重复后形成了紧密相连的mir-9b、mir-9c基因簇,随后经历片段缺失和碱基突变以mir-9a/9、mir-9b的形式存在于节肢动物门和线虫纲等无脊椎动物中.
在由无脊椎动物向脊椎动物进化的过程中mir-9/9a基因可能发生片段重复形成了低等脊椎动物(鱼纲)中的mir-9-1至mir-9-7基因.
2.
4靶基因的预测大量的研究表明,一些miRNA在转录后水平调控脑部基因的表达,而且在神经发育各个阶段都起着重要的调控作用.
miRNA在中枢神经系统的表达具短暂性和区域性[12].
如miR-138和miR-124主要在中枢神经系统中表达,miR-126和miR-29主要在星形胶质细胞中表达.
据报道miR-9是一个组织特异性的miRNA,它主要在中后脑交界处(midbrain-hindbrainboundary,MHB)的邻近区域表达[13],可通过作用于成纤维生长因子(fibroblastgrowthfactor,Fgf)信号通路调控MHB的组织活性,而且miR-9与神经发生、心脏肥大[14]、脑部正常发育[15,16]等具有密切关系.
为进一步了解miR-9参与的调控机制及miRNA作用的新标靶,本文以小鼠的mmu-miR-9基因为研究对象,采用TargetScan、PicTar、microrna.
org和MicroCosmTargrts等四个microRNA靶位点分析软件搜索mmu-miR-9的靶基因.
利用TheGeneOntol-ogy、KyotoEncyclopediaofGenesandGenomes和David软件分析四个靶基因软件均能预测到的靶基因(表3)的功能,从而了解miR-9在生物体中可能的作用路径.
由TheGeneOntology和KyotoEncyclopediaofGenesandGenomes数据库分析可知,miR-9的靶基因多数参与新陈代谢、信号转导、跨膜运输和物质转运等生命活动过程,与神经细胞、心肌细胞等细胞的生长和分化密切相关.
例如:nidogen2所编码的巢蛋白是神经干细胞的标志蛋白,Erbb2ip基因所编码的Erbb2相互作用蛋白是神经干细胞正常生长所必须的表皮生长因子受体(epidermalgrowthfactorreceptor,EGFR),Slc31a2所编码的铜离子转运蛋白对于维持神经系统的正常生理功能非常重要.
可见,miR-9在基因表达调控中具有广泛的作用.
Chun-nian等[17]曾报道miR-9对于神经干细胞的增殖和分化具有调控作用,Krichevsky等[18]在研究胚胎神经干细胞形成时发现以miR-9和miR-124为代表在脑部含量丰富的miRNA,主要是在神经细胞向神经元的分化过程中诱导产生的,而且在神经分化过程中阻断它们的功能会提升神经干细胞向星形胶质细胞分化的可能性.
这些研究工作也支持本文获得的结果.
David数据库的Functionannotationchart的数据统计显示mmu-miR-9的候选靶基因中,与磷酸化和乙酰化作用相关的基因数量最多,暗示着miR-9与表观调控路径相关,为我们研究mir-9的功能提供一个新的切入点.
(续表2)罗丹丹等:miRNA-9基因家族进化分析及其靶基因预测515图1MEGA4.
1软件NJ法构建mir-9基因家族系统进化树缩写含义:bfl:文昌鱼;xtr:爪蟾属;gga:原鸡;cfa:家犬;mdo:短尾猊;lla:绒毛猴属;mml:恒河猴;ggo:大猩猩;has:智人;ptr:黑猩猩属;oan:鸭嘴兽;mmu:小家鼠;bta:欧洲牛;ssc:野猪;dre:斑马鱼;spu:紫色球海胆;sko:Saccoglossuskowalevski;dpu:水蚤属;aga:冈比亚按蚊;dpe:果蝇;dvi:果蝇;lmi:飞蝗;tca:拟谷盗属;cap:Capitellasp.
I;lgi:Lottiagigantean;sme:Schmidteamediterranea.
Fig.
1phylogenetictreeofthemiR-9genefamilyconstructedbyMEGA4.
1softwarebasedonNeighbor-joiningmethodMeaningsofabbreviations:bfl,Branchiostomafloridae;xtr,Xenopustropicalis;gga,Gallusgallus;cfa,Canisfamiliaris;mdo,Monodelphisdomes-tica;lla,Lagothrixlagotricha;mml,Macacamulatta;ggo,Gorillagorilla;has,Homosapiens;ptr,Pantroglodyte;oan,Ornithorhynchusanatinus;mmu,Musmusculus;bta,BosTaurus;ssc,Susscrofa;dre,Daniorerio;spu,Strongylocentrotuspurpuratus;sko,Saccoglossuskowalevski;dpu,Daphniapulex;aga,Anophelesgambiae;dpe,Drosophilapseudoobscura;dvi,Drosophilavirilis;lmi,Locustamigratoria;tca,Triboliumcastaneum;cap,Capitellasp.
I;lgi,Lottiagigantean;sme,Schmidteamediterranea.
516·研究论文·表3四个靶基因分析软件均能预测到的mmu-miR-9靶基因Table3Targetsofmmu-miR-9predictedbyfoursoftwaresNCBI序列号基因名称编码蛋白所处代谢路径及其功能简介Acc.
No.
NameofgenesIntroductionofgenefunctionNM_025286Slc31a2,solutecarrierfamily31,member2Solutetransportfamilyof31,alsoknownascopperiontransportprotein,centralnervoussystemexistenceahighconcentrationofcopperion(Cu2+),andCu2+playaroleinthenerveterminalwhichcandepolarizedfromthesuddenreleasedintothesynapticvesiclescontactgapLaminA,themaincomponentofnuclearlamina,tomaintainlevelsinthenucleusplaysasupportingrole,asthenuclearmembraneandchromatinstructureprovidessupport.
NM_001002011Lmna,laminAMainlyinhypertrophiccardiomyopathy,arrhythmogenicrightventricularNM_011960Parg,poly(ADP-ribose)glycohydrolasecardiomyopathy,cardiomyopathyamplificationpathwaycatalyticactivity,Hydrolyzespoly(ADP-ribose)atglycosidic(1''-2')linkageofribose-ribosebondtoproducefreeADP-ribose.
,function,Poly(ADP-ribose)synthesizedafterDNAdamageisonlypresenttransientlyandisrapidlydegradedbypoly(ADP-riboseglycohydrolase.
Poly(ADP-ribose)metabolismmayberequiredformaintenanceofthenormalfunctionofneuronalcells.
,sequencecaution,TranslatedasMet.
,similarity,Belongstothepoly(ADP-ribose)glycohydrolasefamilyNM_146151Tesk2,testis-specifickinase2Testis-specificproteinkinase2,mainlycontainingN-terminalkinasedomainofserine/threonineproteinkinase,mainlyexpressedintestisandprostateNM_030706Trim2,tripartitemotifprotein2Tripartitemotifprotein2,expressionofihighlevelsinthenervoussystemtomaintainitsstructuralplasticityofneuronsNM_019767Arpc1a,actinrelatedprotein2/3complex,Actinrelatedprotein2/3complexandpromotemicrofilamentformationoflivingcellssurroundingMainlyinvolveinFCgammaR-relatedShijuneffect,adjusttheactincytoskeleton,bacterialmetabolicpathwayssuchasepithelialcellsinfectedNM_199029Zfp395,zincfingerprotein395ZincFingerprotein395,andiscloselyrelatedtoembryonicdevlopmentandcelldifferentiationisanimportantfunctionofaclassoftranscriptionfactorNM_031397Bicc1,bicaudalChomolog1(Drosophila)Two-tail-Cgenehomolog1,playaroleintheheadandtailstructure,theorgandevelopmentNM_011843Mbc2,membraneboundC2domaincontainingMembraneboundC2domaincontainingproteinproteinNM_008695Nid2,nidogen2Nidogen2,oneofembryonicintermediatefilamentprotein,whichisconsideredamarkerofneuralstemcellsNM_021563Erbb2ip,Erbb2interactingproteinErbb2interactingproteinsforepidermalgrowthfactorreceptor(epidermalgrowthfactorreceptor,EGFR)MainlyinvolveinNOD-likereceptorsprimarilysignalpathwayNM_016673Cntfr,ciliaryneurotrophicfactorreceptorCiliaryneurotrophicfactorreceptors,onnervecellgrowth,differentiationhasobviousnutritionalroleMainlyinvolveincytokinesandcytokinereceptorinteractionpathway,Jak-STATsignalingpathwayNM_023245Palmd,palmdelphinPalmdelphin,mainlyinthebrain,thehippocampus,amygdalaNM_134134A630042L21Rik,RIKENcDNAA630042L21geneHighmobilitygroupboxdomaincontainingfamilyofcomplex3NM_013890Fbxw2,F-boxandWD-40domainprotein2F-boxandWD-40domainprotein2NM_009679Ap2m1,adaptorproteincomplexAP-2,mu1AdaptorProteinComplexAP-2,μ1subunit,mainlycatalytic,transportprotein,proteinbinding,lipidbindingMainlyinvolveinendocytosis,Huntingtondiseaseandothermetabolicpathway(待续)罗丹丹等:miRNA-9基因家族进化分析及其靶基因预测517NCBI序列号基因名称编码蛋白所处代谢路径及其功能简介Acc.
No.
NameofgenesIntroductionofgenefunctionNM_134060Slc35b3,solutecarrierfamily35,memberB3Family35solutetransport,membranetransportproteinfamilymember,playsanimportantroleinneuralstemcellrenewalandneurogenesisNM_026139Armcx2,armadillorepeatcontaining,X-linked2Armadillorepeatcontaining,X-linked2,astrongsignalinembryonicdevelopmentstageNM_011945Map3k1,mitogenactivatedproteinkinaseMitogen-activatedproteinkinasekinase1,throughthekinasekinase1MAPKcascadecontrolledresponsesysteminvolvedincellproliferationanddifferentiation,whenthevitalityoutofcontrolwillleadtotumorMainlyinvolveinMAPKsignalpathway,ubiquitin-mediatedproteindegradation,RIG-I-likereceptorsignalingpathway,neurotrophicfactorsignalingpathway,GnRHsignalingpathwayandotherrelatedNM_015736Galnt3,UDP-N-acetyl-alpha-D-galactosamineN-acetylgalactoseaminotransferasepolypeptide,oftenwiththefibroblastgrowthfactor23witheffectMainlyinvolveinO-polysaccharidebiosynthesis,metabolicpathwayNM_009680Ap3b1,adaptor-relatedproteincomplex3,Adaptor-relatedproteincomplex3,beta1subunit,ismainlycatalytic,beta1subunittransportprotein,proteinbinding,lipidbindingMajormetabolicpathwaysassociatedwiththelysosomalNM_146164Lrch4,leucine-richrepeatsandcalponinhomologyLeucine-richrepeatsandcalponinhomology(CH)domaincontaining4,(CH)domaincontaining4embryonicgrowthanddifferentiationofspecificreceptorNM_146087Csnk1a1,caseinkinase1,alpha1Caseinkinase1,intheWntpathwayplaysanimportantroleinMainlyinvolveinWntsignalingpathway,HedgehogsignalingpathwayNM_023670Igf2bp3,insulin-likegrowthfactor2,bindingInsulin-likegrowthfactor2,bindingprotein3protein3NM_1786134933433P14Rik,UPF0279proteinRIKENcDNNA4933433P14geneC14orf129homolog.
NM_133195Brunol4,bruno-like4,RNAbindingproteinBruno-like-Gene4,RNAbindingprotein,playaroleinalternativesplicingandthetargetmRNAtranslationandstability3讨论microRNA作为重要的内源性调控基因,分布范围广,参与的生物学过程复杂,调控靶基因数众多,对于生物的正常生长发育至关重要,在动植物的进化过程中扮演着重要的角色.
mir-9基因家族在原口动物和后口动物中分布广泛,且高度保守,说明mir-9基因在生物起源和迁移过程中的重要作用.
mir-9基因家族成熟序列的2-8位碱基即"种子区域"的保守性为100%,暗示其功能的保守性.
从低等向高等进化过程中普遍存在mir-9基因家族的调控机制,说明miRNA的调控机制对物种生存的重要性[19].
Maller等[20]在通过分析拟南芥基因组,发现许多miRNAs家族是通过基因组水平上的复制、串联重复或者大片段复制产生的,接着再发生时空表达的分化.
Tanzer等[21]发现miR-17基因簇进化历史伴随着串联重复、整个基因簇复制以及个别miRNA的随机缺失,而且miR-17基因家族可能与脊椎动物的早期进化有关.
对于mir-9基因的进化分析显示,mir-9、mir-9b、mir-9c、mir-9a基因主要出现在进化早期,为祖先基因,且mir-9b和mir-9c聚集成簇.
Hertel等[22]在后生动物基因组中的miRNA基因家族之间的系统发育分析揭示了串联重复和片段重复是miRNA基因簇进化的主要机制,也因此产生了miRNA基因簇和miRNA基因家族的多样性分布.
由此推测miR-9b和miR-9c两个基因在同一染色体上所形成的基因簇可能是由串联重复所产生的.
mir-9-1至mir-9-7则主要分布在两栖类及哺乳类等较高等的脊椎动物中,mir-9基因家族从无脊椎动物向脊椎动物进化的过程中数量有所增多,可能发生了串联重复、大片段重复和个别miRNA的缺失.
所以对mir-9的系统进化分析不仅有利于揭示非编码小分子RNA的起源及其作用机制,还可以增进对物种进(续表3)518·研究论文·化的了解.
Kosik等[23]和Landgraf等[24]对于microRNA的研究显示,miRNA在神经系统中含量丰富,表达具有组织特异性和非对称性,说明miRNAs在神经系统的特定功能中所扮演的重要角色.
Monika等[5]利用原位杂交技术发现miR-124及miR-9在神经系统中表达丰富,miR-9主要在神经前体细胞和神经元中表达,它是神经发育和成脑中所出现的特异性miRNA[25,26].
本文利用生物信息学方法,在四个miRNA在线分析软件中搜索miR-9的共同靶基因,通过GO和KEGG分析显示mir-9基因家族可通过其靶基因作用于多条代谢路径,且与神经系统正常发育与生长途径具有密切关系.
所以对于mir-9基因家族的靶基因的预测不仅有利于了解其可能参与的生物学途径,还可为以后的实验研究提供理论依据.
参考文献(References)1DennisC.
ThebravenewworldofRNA.
Nature2002;418(6894):122-4.
2RuvkunG.
Molecularbiology,glimpsesofatinyRNAworld.
Science2001;294(5543):797-9.
3LauNC,LimLP,WeinsteinEG,BartelDP.
AnabundantclassoftinyRNAswithprobableregulatoryrolesinCaenorhabditiselegans.
Science2001;294(5543):858-62.
4RodriguezA,Griffiths-JonesS,AshurstJL,BradleyA.
Identi-ficationofmammalianmicroRNAhostgenesandtranscriptionunits.
GenomeRes2004;14(10A):1902-10.
5DeoM,YuJY,ChungKH,TippensM,TurnerDL.
DetectionofmammalianmicroRNAexpressionbyinsituhybridizationwithRNAoligonucleotides.
DevDyn2006;235(9):2538-48.
6YooAS,StaahlBT,ChenL,CrabtreeGR.
MicroRNA-mediatedswitchingofchromatin-remodellingcomplexesinneuraldevel-opment.
Nature2009;460(7255):642-6.
7DelaloyC,LiuL,LeeJA,SuH,ShenF,YangGY,etal.
Mi-croRNA-9coordinatesproliferationandmigrationofhumanembryonicstemcell-derivedneuralprogenitors.
CellStemCell2010;6(4):323-35.
8MaL,YoungJ,PrabhalaH,PanE,MestdaghP,MuthD,etal.
miR-9,aMYC/MYCN-activatedmicroRNA,regulatesE-cadher-inandcancermetastasis.
NatCellBiol2010;12(3):247-56.
9TavazoieSF,AlarcónC,OskarssonT,PaduaD,WangQ,BosPD,etal.
EndogenoushumanmicroRNAsthatsuppressbreastcancermetastasis.
Nature2008;451(7175):147-52.
10DoenchJG,SharpPA.
SpecificityofmicroRNAtargetselectionintranslationalrepression.
GenesDev2004;18(5):504-11.
11SaitouN,NeiM.
Theneighbor-joiningmethod:anewmethodforreconstructingphylogenetictrees.
MolBiolEvol1987;4:406-25.
12WangJF,ZhouH,ChenYQ,LuoQJ,QuLH.
Identificationof20microRNAsfromOryzasativa.
NucleicAcidsRes2004;32(5):1688-95.
13DelaloyC,GaoFB.
microRNA-9multitaskingnearorganizingcenters.
NatNeurosci2008;11:625-6.
14WangK,LongB,ZhouJ,LiPF.
miR-9andNFATc3regulatemyocardinincardiachypertrophy.
JBiolChem2010;285(16):11903-12.
15ZhangZ,ChangH,LiY,ZhangT,ZouJ,ZhengX,etal.
Mi-croRNAs:potentialregulatorsinvolvedinhumananencephaly.
IntJBiochemCellBiol2010;42(2):367-74.
16CoolenM,Bally-CuifL.
MicroRNAsinbraindevelopmentandphysiology.
CurrOpinNeurobiol2009;19(5):461-70.
17ZhaoC,SunG,LiS,ShiY.
AfeedbackregulatoryloopinvolvingmicroRNA-9andnuclearreceptorTLXinneuralstemcellfatedetermination.
NatStructMolBiol2009;16(4):365-71.
18KrichevskyAM,SonntagKC,IsacsonO,KosikKS.
SpecificmicroRNAsmodulateembryonicstemcell-derivedneurogenesis.
StemCells2006;24:857-64.
19MassirerKB,PasquinelliAE.
TheevolvingroleofmicroRNAsinanimalgeneexpression.
Bioessays2006;28:449-52.
20MaherC,SteinL,WareD.
EvolutionofArabidopsismicroRNAfamiliesthroughduplicationevents.
GenomeRes2006;16:510-9.
21TanzerA,StadlerPF.
MolecularevolutionofamicroRNAclus-ter.
JMolBiol2004;339:327-35.
22HertelJ,LindemeyerM,MissalK,FriedC,TanzerA,FlammC,etal.
TheexpansionofthemetazoanmicroRNArepertoire.
BMCGenomics2006;7:25.
23KosikKS.
TheneuronalmicroRNAsystem.
NatRevNeurosci2006;7(12):911-20.
24LandgrafP,RusuM,SheridanR,SewerA,IovinoN,AravinA,etal.
AmammalianmicroRNAexpressionatlasbasedonsmallRNAlibrarysequencing.
Cell2007;129:1401-14.
25Lagos-QuintanaM,RauhutR,YalcinA,MeyerJ,LendeckelW,TuschlT.
Identificationoftissue-specificmicroRNAsfrommouse.
CurrBiol2002;12:735-9.
26KrichevskyAM,KingKS,DonahueCP,KhrapkoK,KosikKS.
AmicroRNAarrayrevealsextensiveregulationofmicroRNAsduringbraindevelopment.
RNA2003;9(10):1274-81.
罗丹丹等:miRNA-9基因家族进化分析及其靶基因预测519MolecularEvolutionofmiRNA-9GeneFamilyandPredictionofTheirTargetGenesDan-DanLuo,Yu-PingLuo,JuanPeng,Si-GuangLi*(CollegeofLifeSciencesandFoodEngineering,NanchangUniversity,Nanchang330031,China)AbstractmicroRNAsareaclassofnon-codingsmallRNAsof21~25ntsineucaryota,whichregulategeneexpressionatthepost-transcriptionallevel.
Inthisstudy,wesearchedmir-9genesofmetazoainmiRBase,andgotatotalof120sequencesin47specieswhichindicatingtheextensiveexistenceofmir-9geneindifferentspecies.
Theanalysisofgenelocalizationshown86%ofmir-9geneslocateintheintergenicregion(IGR).
Multiplesequencealignmentofmaturesequencesofmir-9genefamilyshowedthatthesecondtoeighthbasesandthefour-teenthtoeighteenthbasesareconservativebases.
Phylogeneticanalysisrevealedmir-9bandmir-9cmaybetheear-liestgeneformsofthisfamily,viz.
ancestralgenesofmir-9family.
Theseancestralgenescreatedmir-9-1tomir-9-7genesinvertebratebytandemduplication,largersegmentduplication,deletionandmutationofindividualbases.
Targetgenesofmmu-miR-9arepredictedbyfourmiRNAtargetgenepredictionsoftwares.
TheresultshowedthatmiR-9wasinvolvedinnervoussystemdevelopment,cardiacdiseasesandtransmembranetransportsystem.
TheseresultscontributetofurtherstudyontheroleofmicroRNAinnervoussystemdevelopmentandthemechanismofneuralcellgrowthanddifferentiation.
KeywordsmiRNA-9;evolution;targetgeneReceived:October11,2011Accepted:February25,2011ThisworkwassupportedbytheNationalNaturalScienceFoundationofChina(No.
30971473,No.
31071138)*Correspondingauthor.
Tel:86-791-8304099,E-mail:siguangli@163.
com
- 基因nod32相关文档
- 帐户nod32
- 设置[笔记]ESET NOD32如何升级与设置
- 私服地址NOD32免ID升级服务器
- 设置[教学]ESET NOD32如何升级与设置
- 服务器NOD32升级服务器制作
- 测量【精选】两种方法架设NOD32升级服务器
提速啦:美国多IP站群云服务器 8核8G 10M带宽 7IP 88元/月
提速啦(www.tisula.com)是赣州王成璟网络科技有限公司旗下云服务器品牌,目前拥有在籍员工40人左右,社保在籍员工30人+,是正规的国内拥有IDC ICP ISP CDN 云牌照资质商家,2018-2021年连续4年获得CTG机房顶级金牌代理商荣誉 2021年赣州市于都县创业大赛三等奖,2020年于都电子商务示范企业,2021年于都县电子商务融合推广大使。资源优势介绍:Ceranetwo...
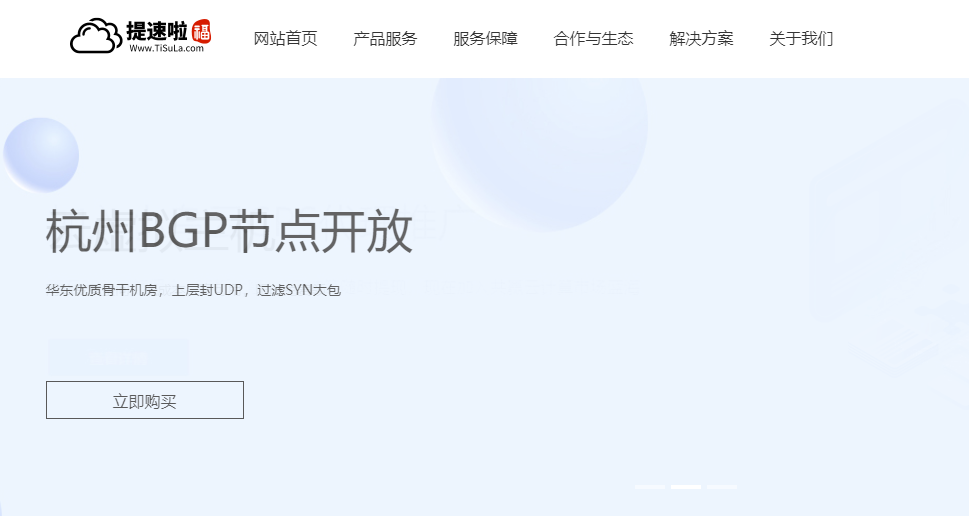
恒创新客(317元)香港云服务器 2M带宽 三网CN2线路直连
恒创科技也有暑期的活动,其中香港服务器也有一定折扣,当然是针对新用户的,如果我们还没有注册过或者可以有办法注册到新用户的,可以买他们家的香港服务器活动价格,2M带宽香港云服务器317元。对于一般用途还是够用的。 活动链接:恒创暑期活动爆款活动均是针对新用户的。1、云服务器仅限首次购买恒创科技产品的新用户。1 核 1G 实例规格,单个账户限购 1台;其他活动机型,单个账户限购 3 台(必须在一个订单...
ZJI韩国BGP+CN2服务器,440元起
ZJI又上新了!商家是原Wordpress圈知名主机商:维翔主机,成立于2011年,2018年9月启用新域名ZJI,提供中国香港、台湾、日本、美国独立服务器(自营/数据中心直营)租用及VDS、虚拟主机空间、域名注册等业务。本次商家新上韩国BGP+CN2线路服务器,国内三网访问速度优秀,适用8折优惠码,优惠后韩国服务器最低每月440元起。韩国一型CPU:Intel 2×E5-2620 十二核二十四线...
nod32 升级为你推荐
-
网站运营网络运营具体做什么呢天天酷跑刷金币天天酷跑怎么刷金币?godaddy通过什么网址可以查godaddy的域名信息奇虎论坛奇虎论坛最新推荐歌曲列表·免费免费建站我想建一个自己的免费网站,但不知道那里有..网站优化方案网站优化方法有哪些blogcn南京明城墙(太平门一带某些地区)的城砖上为什么会有一些小洞(每块砖两个洞洞……)?如何清理ie缓存怎么样清理IE缓存?网络虚拟机虚拟机的网络怎么弄?php购物车PHP中用json实现购物车功能,怎么实现